Dr. Theodore Roth
Marson研究室(University of California San Francisco)
遺伝子編集実験で特に必要とされる高濃度の長鎖ssDNA配列は、研究室での合成が難しいのですが、GenScriptの長鎖ssDNAを使用して、ヒトプライマリーT細胞への長鎖DNA配列導入に成功できました。
ホーム » CRISPRサービス » HDRノックイン用テンプレート » GenExact™ 一本鎖DNA合成サービス
一本鎖DNA(ssDNAまたはssODN)は、高い編集効率かつオフターゲットを減少した、遺伝子ノックイン作製のための最良なCRISPR相同組換え修復(HDR)テンプレートとして、皆様にお使いいただいています。GenScriptは、お客様のCRISPR実験の編集効率を最大化するために、高品質で配列検証済のssDNAを提供しています。

Dr. Theodore Roth
Marson研究室(University of California San Francisco)
遺伝子編集実験で特に必要とされる高濃度の長鎖ssDNA配列は、研究室での合成が難しいのですが、GenScriptの長鎖ssDNAを使用して、ヒトプライマリーT細胞への長鎖DNA配列導入に成功できました。
ssDNA
最速3週間でお届け
| Quantity (ug) | <500nt (JPY/Item) | 501-5000nt (JPY/Item) |
|---|---|---|
| 3 | ¥64,000 | ¥64,088~¥472,000 |
| 5 | ¥80,000 | ¥80,096~¥552,000 |
| 10 | ¥104,000 | ¥104,104~¥624,000 |
| 20 | ¥120,000 | ¥120,112~¥704,000 |
| 50 | ¥192,000 | ¥192,128~¥984,000 |
| 100 | ¥240,000 | ¥240,136~¥121,6000 |
| >100 | お問い合わせください | |
5 kbより長い配列は、 [email protected]までお問い合わせください。
*液体製品を納品する場合に、追加送料があります。
| 試験内容 | 試験方法 | 合格基準 | リサーチグレード (≤2mg) |
|---|---|---|---|
| 純度 | アガロースゲル電気泳動 | 単一バンド | ✔ |
| シークエンスの正確性 | サンガーシークエンス | 100%正確であること | ✔ |
| 吸光度 | 分光光度計 260 nm/230 nm | ≥ 2.0 | ✔ |
CRISPRベースの遺伝子挿入、置換および修正
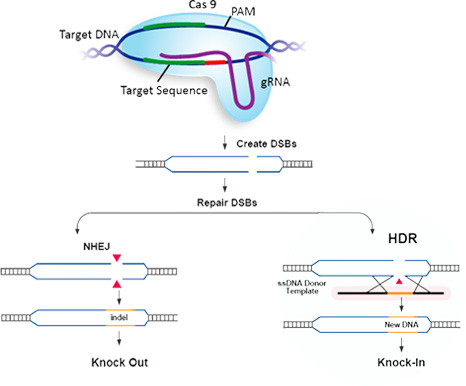
HDRベースのCRISPR遺伝子編集のメカニズム
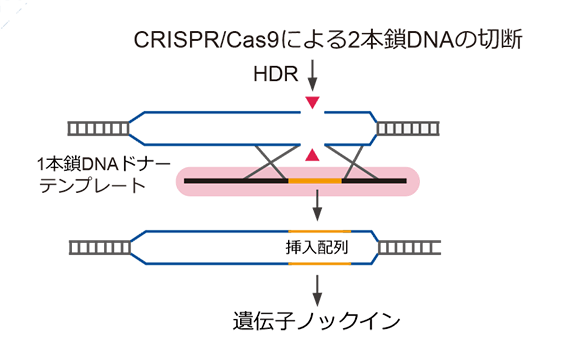
CRISPR/Cas9は、ターゲットDNA部位へ正確に二本鎖切断(DSBs)を行うため、広く普及しています。ガイドRNA(gRNA)はCas9タンパク質と複合体を形成した後、ターゲットDNA上のprotospacer adjacent motif(PAM)配列を認識します。Cas9は、エンドヌクレアーゼ活性によりDSBsを引き起こし、その後2種類の修復機構を誘導します。一つは非相同末端結合(NHEJ: non-homologous end-joining)で、DSB部位に変異を導入します。もう一方のメカニズムは相同組換え修復(HDR: homology directed repair)で、切断部位にドナーDNAを挿入し、遺伝子ノックインを可能にします。従来、HDR用のドナーDNAテンプレートとして、二本鎖DNA(dsDNA)使用されてきましたが、最近の研究では、一本鎖DNA(ssDNAまたはssODN)がCRISPRベースの遺伝子挿入、置換及び修正で最適なHDRテンプレートであることが示されました1-5。
関連文献
二本鎖DNAドナーと比較して、一本鎖DNAでは、特にプライマリー細胞、幹細胞およびトランスジェニック動物モデルの開発において、ゲノム編集効率や特異性の向上、およびオフターゲットの減少が証明されています1-5。
実験目的、および宿主細胞株によります。二本鎖DNAドナーと比較して、一本鎖DNAでは、特にプライマリー細胞、幹細胞およびトランスジェニック動物モデルの開発において、ゲノム編集効率や特異性の向上、およびオフターゲットの減少が証明されています。
| Plasmid DNA | dsDNA | ssDNA | |
|---|---|---|---|
| HDR efficiency | Medium | High | Comparable to dsDNA |
| Off-target rate | Medium | High | Low |
| Cost | Lower | Medium | High |
| Toxicity | High | High | Low |
点変異には非対称ssDNAのデザインを使用することが提唱されています。その他のデザインの秘訣は、こちらの論文をご覧ください。長鎖遺伝子の挿入は、挿入遺伝子に300~1000 bpのホモロジーアームを付与したものが報告されています。多くの場合、500 bpのホモロジーアームが使用されています。二回目以降の切断を避けるため、ssDNAテンプレートはPAM配列にサイレント変異を含むようデザインすることが重要です。ノックインドナーデザインは複雑になることがあります。ソフトウェアを使用して(例.snapgene)、配列を表示・編集することを強く推奨します。
挿入遺伝子に300~1000 bpのホモロジーアームを付与したものが報告されています。多くの場合、500 bpのホモロジーアームが使用されています。
マウスにおいてCRISPR/Cas9を介したヌルアレル作成は非常に効率的ですが、コンディショナルアレル作成はより難しいことが証明されています。
問題の特定と解決のため、以下のワークフローを参照してください。
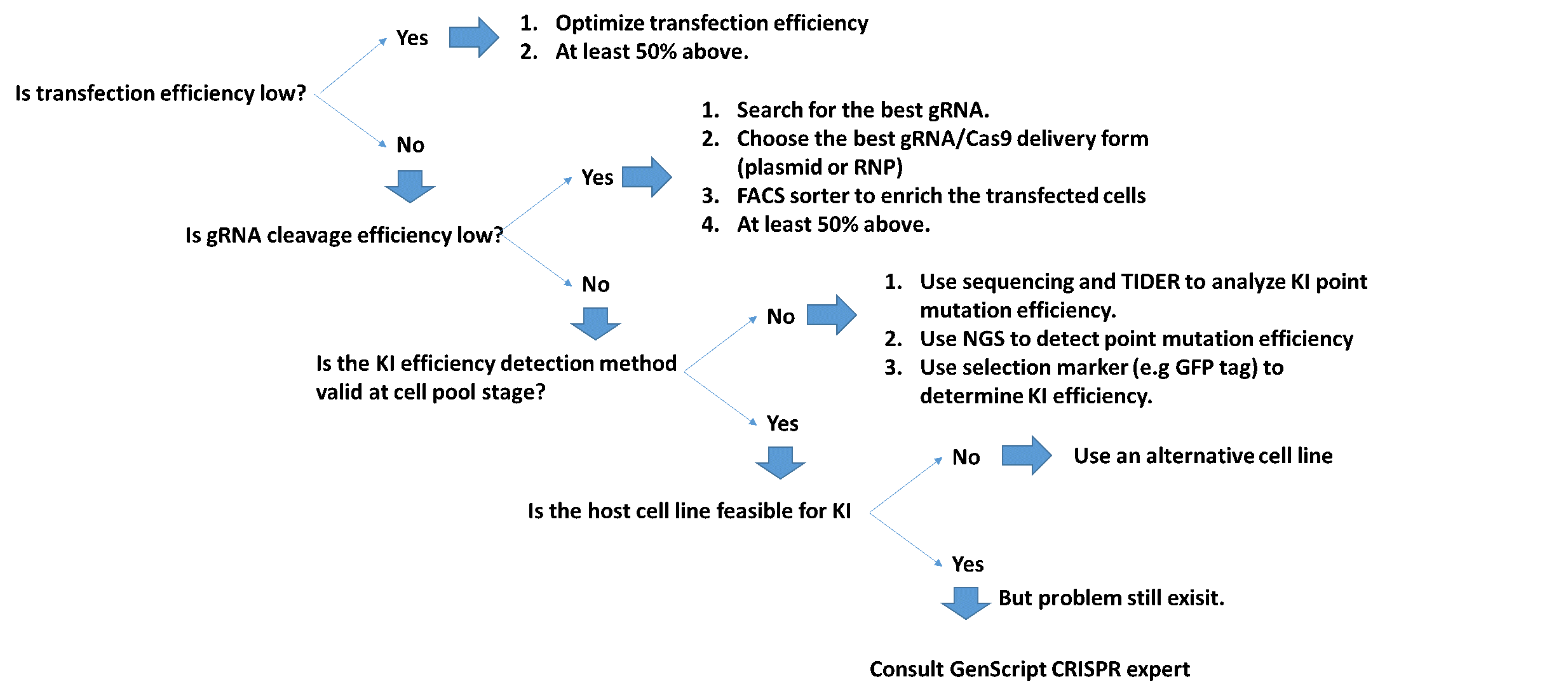
GenScriptでは、3000 nt未満の長さの配列は日常的に作成しています。当社は過去に、GC含量とリピート配列が非常に多いモチーフを持つ最大5000 ntのssDNA合成に成功しています。3000 ntよりも長いssDNAの合成をご希望の場合、当社の技術サポートチームまでご相談ください。
GenScriptがお届けする最大量は、配列の長さが< 1 kbの場合は40 µg、1-3 kbの場合は20 µg、>3 kbの場合は最大収量が5 µgとなります。
当社は凍結乾燥前の最終ssDNA産物に、二つの主な品質管理試験を実施します: 1) ssDNAの配列を保証するためのサンガーシークエンシング; 2) ゲル電気泳動による最終ssDNA産物の純度試験。
ssDNAの生産中、当社では配列精度の保証のため、二回のシークエンシングを行います。まず最初に、プラスミドDNAテンプレートに対してシークエンシングを行い、配列に間違いがないか検証します。さらに、最終ssDNA産物に対してもダイレクトシークエンシングを行い、ssDNA製品の同一性を確認しています。
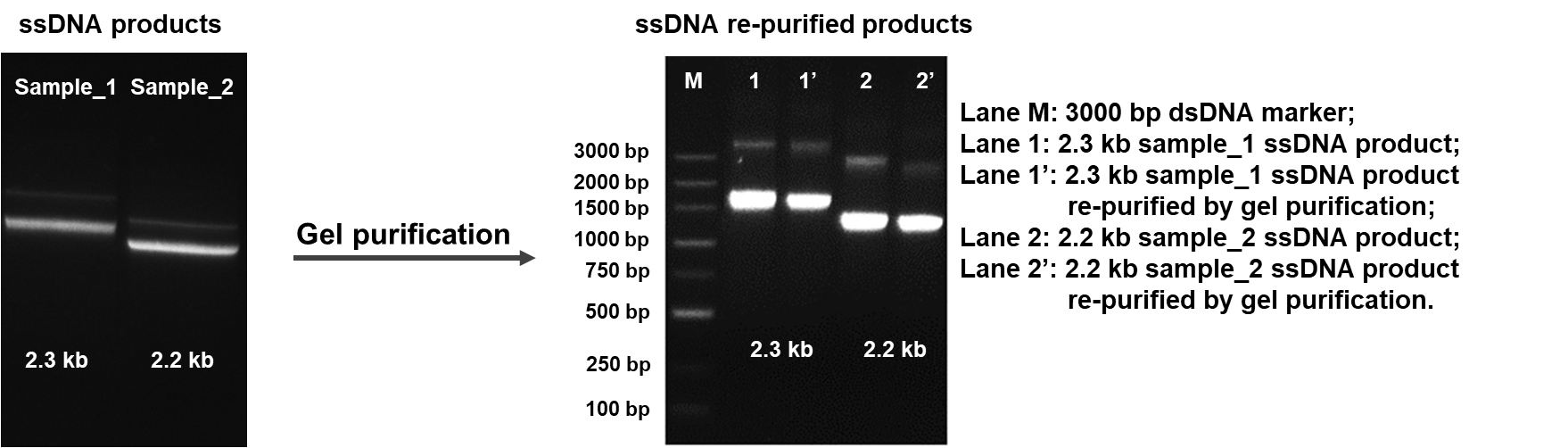
高収率かつ高純度、および化学物質の混入を防ぐため、当社は磁気ビーズとアガロースゲル精製の利点を組み合わせた方法を用いています。
必要ありません。最終ssDNA産物は、配列検証済のssDNA分子のみを含んでいます。GenScriptは、当社独自・特許取得済みの合成酵素アプローチにより、当社のssDNA製品はdsDNAが検出できないレベルであることを保証しています。
いくつかのssDNA産物は、その本来のヌクレオチド配列により、強い凝集傾向を持つ事があります。これらの凝集は分子内および/または分子間力によって形成されることがあります。
ゲル画像内の余分なバンドがssDNAの凝集またはdsDNAの混入かを試験するため、dsDNAは分解せずにssDNAを分解する、S1ヌクレアーゼを使用した消化試験の追加可能です。ssDNA産物が100%の純度であればdsDNAが含まれないため、全てのサンプルはS1ヌクレアーゼで消化され、ゲル電気泳動を行った後にバンドが確認されなくなります。逆に、ssDNA産物にdsDNAが混入している場合、S1ヌクレアーゼの添加後も完全には消化されず、ゲル画像上にバンドが現れたままとなります。

追加実験として、他のバンドがssDNA凝集物であるか確認するために、ゲル上のssDNAのバンドを切り出し、二回目のゲル電気泳動を実施しすることも可能です。目的サイズ以外のバンドがssDNA分子の凝集物である場合、精製後のゲル電気泳動でも現れてきます。さらに、ssDNAバンドの比率は、二つのゲルの間で同様のままとなります。
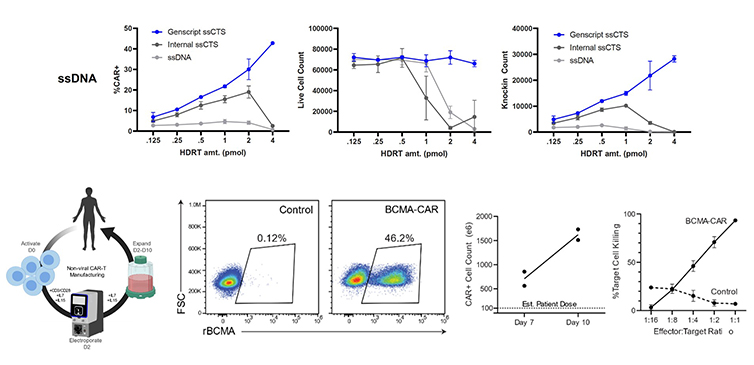
UCSF(University of California San Francisco)のMarson研究室との共同研究で、GenExact™ ssDNAは研究室内で生成したHDRTs(相同組換え修復テンプレート)を一貫して上回り、より低レベルの毒性と高いノックイン効率を示しました。エレクトロポレーションによる、エンハンサーを使用しないGenExact™ ssDNAのノックイン効率は、GMP互換スケールで46.2%に達しました。GenExact™ ssDNAで生成されたノックイン陽性細胞の数は、想定患者投与量(100*10e6)をはるかに上回りました。In vitroアッセイではBCMA-CAR細胞による効率的な細胞死が証明されました。
Brian R Shy. et.al., bioRxiv(2021), Read the whole article.
![]() CRISPR Knock-in Protocol for HEK 293T/293 cells with Thermo Fisher Neon®
Transfection System
CRISPR Knock-in Protocol for HEK 293T/293 cells with Thermo Fisher Neon®
Transfection System
![]() CRISPR Knock-in Protocol for Jurkat cells with Thermo Fisher Neon® Transfection
System
CRISPR Knock-in Protocol for Jurkat cells with Thermo Fisher Neon® Transfection
System
![]() CRISPR Knock-in Protocol for stimulated Human T Cells with Lonza 4D Nucleofector™
X Unit
CRISPR Knock-in Protocol for stimulated Human T Cells with Lonza 4D Nucleofector™
X Unit
![]() CRISPR Knock-in Protocol for stimulated Human T Cells with Maxcyte® Electroporation System
CRISPR Knock-in Protocol for stimulated Human T Cells with Maxcyte® Electroporation System

Non-viral HDR Templates Flyer
無料ダウンロード
